Fagdisplay
Fagdisplay eller fagpresentation är en bioteknisk metod för att belysa interaktioner protein–protein, protein–peptid, och protein–DNA, för utveckling av nya biologiska läkemedel och för sökandet efter specifika antikroppar för terapeutiska, diagnostiska eller experimentella tillämpningar av stor betydelse.
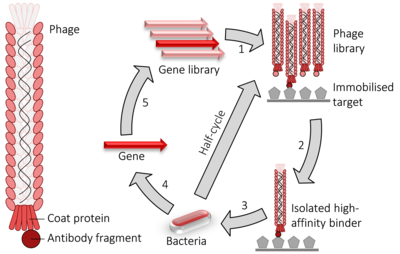
Med genteknologi injiceras en gen som kodar för proteinet av intresse, in i en bakteriofags arvsmassa, så att proteinet presenteras på utsidan av fagen vilket möjliggör isolering och identifikation av ligander. Fagen visar så ett sammanhang mellan genotyp och fenotyp och kan isoleras med hjälp av proteinets bindningsspecificitet. Den isolerade fagen är därefter utgångspunkt för isolering och användning av genen för proteinet av intresse: antikropp, enzym och annat. Se en schematisk illustration av tekniken här.[1]
Nobelpriset i kemi 2018 delades ut till George P. Smith och Gregory P. Winter för deras arbete med utveckling och användning av fagdisplay.[2][3]
Källor
redigera- ^ Phage display, illustration. Nobelprize.org
- ^ Press release: The Nobel Prize in Chemistry 2018
- ^ Smith GP (June 1985). ”Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface”. Science (4705): sid. 1315–7. doi:.